10.4 Explicación de predicción
Una crítica que se hace a los modelos de Machine Learning es que están muy orientados hacia la predicción lo que provoca que sean modelos muy complejos convirtiéndose en cajas negras y de los que perdemos la “explicatividad” del modelo. Esto conlleva que los usuarios finales para los que se desarrollan los modelos puedan perder el interés por el ya que no entienden la naturaleza de las predicciones. Esto es especialmente relevante en entornos médicos: si se construye un modelo para determinar si se debe hacer una intervención, para que el equipo médico pueda confiar en el modelo necesita entender qué soporta la decisión de intervenir al margen de indicadores formales.
Para solventar estos problemas surge una iniciativa relativamente novedosa conocida como LIME. La referencia se puede consultar en el siguiente paper: [https://arxiv.org/pdf/1602.04938v1.pdf].
La idea intuitiva que hay detrás de esta técnica es que, aunque un modelo pueda ser altamente complejo y flexible, se supone que es localmente estable:

Por ejemplo, si entrenasemos una red neuronal para intenar describir los elementos de una fotografía, nos interesaría saber qué es lo que soporta la decisión

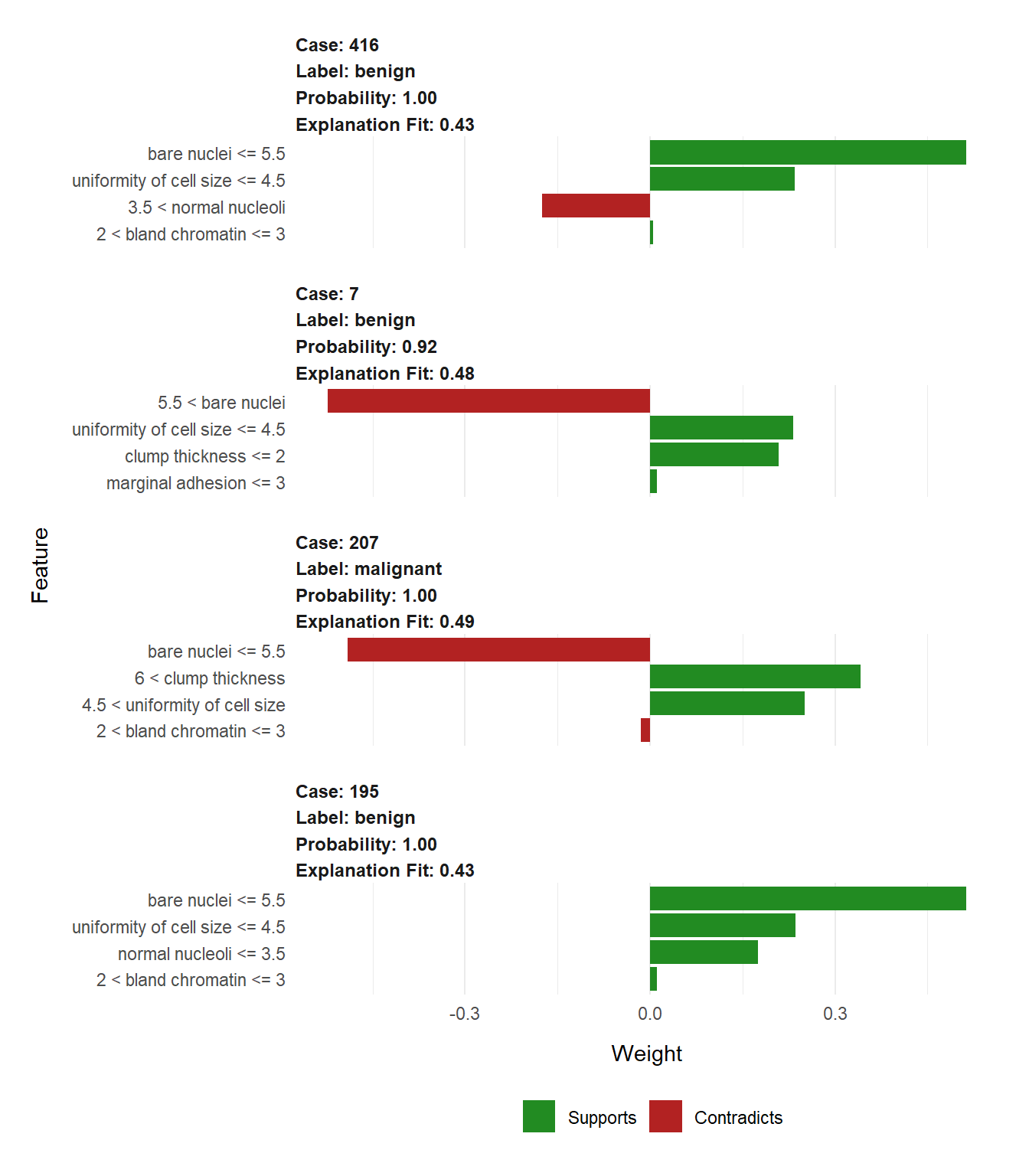
10.4.1 Un ejemplo en R
library(lime)
library(MASS)
data(biopsy)
biopsy$ID <- NULL
biopsy <- na.omit(biopsy)
names(biopsy) <- c('clump thickness', 'uniformity of cell size',
'uniformity of cell shape', 'marginal adhesion',
'single epithelial cell size', 'bare nuclei',
'bland chromatin', 'normal nucleoli', 'mitoses',
'class')
set.seed(4)
test_set <- sample(seq_len(nrow(biopsy)), 100)
prediction <- biopsy$class
biopsy$class <- NULL
model <- lda(biopsy[-test_set, ], prediction[-test_set])
explainer <- lime(biopsy[-test_set,],
model)
explanation <- explain(biopsy[test_set[1:4], ], explainer, n_labels = 1, n_features = 4)
plot_features(explanation, ncol = 1)